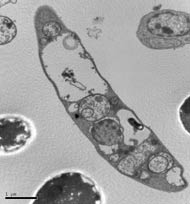
O ministro da Ciência e Tecnologia, Ronaldo Sardenberg, anunciou, em dezembro de 2001, a conclusão do sequenciamento do DNA da bactéria Chromobacterium violaceum, microrganismo de grande potencial medicinal, ecológico e industrial. “O Brasil é o sexto país no mundo a fazer isso”, disse o ministro.
O ministro da Ciência e Tecnologia, Ronaldo Sardenberg, anunciou, em dezembro de 2001, a conclusão do sequenciamento do DNA da bactéria Chromobacterium violaceum, microrganismo de grande potencial medicinal, ecológico e industrial. “O Brasil é o sexto país no mundo a fazer isso”, disse o ministro.
A descrição do DNA da bactéria representa a expansão de fronteiras da genômica no Brasil e a consolidação de um bem-sucedido modelo de trabalho cooperativo. A iniciativa foi feita à imagem do sequenciamento da bactéria Xylella fastidiosa, realizado por 35 laboratórios paulistas e concluído em 2000. O princípio era formar pesquisadores e promover a cooperação entre grupos de pesquisa fisicamente distantes. Essa pesquisa se realiza há mais de um ano com a participação de 25 laboratórios e 200 cientistas, integrantes da Rede Nacional do Projeto Genoma Brasileiro. A conclusão do sequenciamento não implica o desmantelamento da rede. Além de dar continuidade à investigação da Chromobacterium, os cientistas do Projeto Genoma Brasileiro já anunciaram que pretendem repetir a fórmula de sucesso e vão sequenciar em 2002 o genoma de outro microrganismo de importância estratégica. O projeto deve contar mais uma vez com o financiamento do CNPq. Ainda falta definir o organismo a ser sequenciado.
Em outubro de 2000, o CNPq começou a organizar a rede de grupos de pesquisa que participariam da empreitada. Após a análise das 72 propostas recebidas, os 25 laboratórios mais capacitados foram selecionados para compor a Rede Nacional do Projeto Genoma Brasileiro. Os eleitos estão distribuídos por 15 estados das cinco regiões do país. Assim como no caso da Xylella fastidiosa, foi escolhida uma bactéria de interesse econômico. A coordenação do sequenciamento ficou a cargo do bioquímico Andrew Simpson, do Instituto Ludwig de Pesquisa sobre o Câncer, cuja sede brasileira é situada em São Paulo. Para gerenciar o andamento dos trabalhos e tomar as decisões de caráter coletivo, Simpson visitou a maioria dos laboratórios envolvidos. “Comandar o trabalho de grupos de pesquisa espalhados por todo o Brasil foi um desafio maior e mais difícil do que desenvolver o caso da rede Onsa. Mas foi um grande prazer realizar essa tarefa”, observa o bioquímico. Andrew Simpson participou ainda da capacitação dos cerca de 200 pesquisadores envolvidos no projeto. Junto a ele, orientaram também o treinamento dos participantes a bióloga Ana Tereza Ribeiro de Vasconcelos, que coordena, no Laboratório Nacional de Computação Científica do Ministério da Ciência e Tecnologia (LNCC/MCT), o conjunto das informações produzidas pela rede, e o biologista molecular Jesus A. Ferro, da Universidade Estadual Paulista (UNESP) de Jaboticabal, responsável pela organização das bibliotecas de DNA, a coleção de fragmentos com o genoma da Chromobacterium violaceum a serem sequenciados.
Para garantir a agilidade da cooperação entre grupos de pesquisa situados, às vezes, a milhares de quilômetros de distância uns dos outros, foi preciso estabelecer uma rígida dinâmica de trabalho. Os fragmentos de DNA foram distribuídos entre os 25 laboratórios, que realizaram o sequenciamento separadamente. Os dados obtidos nessa etapa seguiram para o Laboratório de Bioinformática do LNCC, que centralizou e processou o conjunto das informações geradas pelo esforço nacional. “Embora alguns laboratórios tenham se destacado na tarefa de sequenciamento, todos eles, uns com mais, outros com menos dificuldade, conseguiram cruzar a linha de chegada”, comemora Ana Tereza. Segundo a pesquisadora do LNCC, alguns grupos do Nordeste e do Norte partiram praticamente do zero, superaram inúmeros desafios e acabaram dando conta do recado. Ao cabo de um ano, estava cumprida a meta inicial: criar uma massa de pesquisadores em todo o país capacitados para o sequenciamento de genomas. Embora ambiciosa e algo arriscada, ela foi plenamente atingida. “O que antes estava restrito a laboratórios da região centro-sul do país rapidamente se espalhou de Manaus a Porto Alegre”, afirma Ana Tereza.
A Chromobacterium violaceum, escolhida para inaugurar as atividades da Rede Nacional do Projeto Genoma Brasileiro, já era conhecida há tempos pelos cientistas por sua capacidade de combater doenças como o Mal de Chagas e a leishmaniose, produzir plásticos biodegradáveis ou reduzir impactos da poluição em áreas de garimpo. Pesquisas recentes apontam ainda sua eficiência no controle de pragas agrícolas. Tudo isso justifica o esforço feito para desvendar seu material genético. Afinal, esse é o primeiro passo para se entender como os genes conferem ao microrganismo tantas e tão preciosas propriedades. Ao criar a Rede Nacional do Projeto Genoma, o CNPq selecionou não só os laboratórios que a integrariam, mas também o organismo que ela iria sequenciar. Venceu a proposta apresentada pelo grupo da bióloga Tânia Creczynski-Pasa, do Departamento de Ciências Fisiológicas da Universidade Federal de Santa Catarina (UFSC), em Florianópolis.
A produção de violaceína, vale destacar, é uma das características mais notáveis da Chromobacterium. Além de possuir ação antibiótica, esse pigmento de cor escura se mostrou eficaz também no combate ao Trypanosoma cruzi, causador do Mal de Chagas, e no tratamento da leishmaniose, doença provocada por protozoários do gênero Leishmania. Suspeitas sobre a ação antibiótica da violaceína foram levantadas a partir da observação da microfauna do Rio Negro, na Amazônia, onde a Chromobacterium violaceum está presente em grande quantidade. Boa parte dos pequenos animais de suas águas, verificou-se depois, é dizimada em virtude do poder antibiótico da substância. Diante da baixa disponibilidade de alimentos, os cardumes desaparecem e tornam inviáveis as atividades pesqueiras da população ribeirinha. Não é por outra razão que o rio ficou conhecido internacionalmente como “hungry river” (rio da fome).
Em artigo publicado na revista Ciência Hoje em 1990, o pesquisador Luiz Renato Caldas, do Instituto de Biofísica da Universidade Federal do Rio de Janeiro, afirma que a biomassa do Rio Negro chega a ser 200 vezes menor que a do Rio Amazonas. A Chromobacterium não é encontrada exclusivamente na região do Rio Negro, como é costume imaginar. Ela se difunde amplamente por todo o mundo tropical e não apenas em ambientes aquáticos (algumas cepas já foram isoladas em amostras de solo). Além de violaceína, a Chromobacterium produz também um peptídeo substância formada pela união de dois ou mais aminoácidos, como as proteínas e certos hormônios, que revelou expressiva atividade antitumoral em testes feitos com camundongos.
A Chromobacterium é um dos raros microrganismos já estudados até agora que sintetizam polihidroxialcanoatos (PHAs) polímeros orgânicos com características físico-químicas muito semelhantes às do polipropileno e do polietileno. “Isso significa que os PHAs são uma excelente alternativa aos plásticos derivados da indústria petroquímica”, traduz o biólogo Fabrício Rodrigues dos Santos, do Laboratório de Biodiversidade e Evolução Molecular da Universidade Federal de Minas Gerais, em Belo Horizonte, um dos 25 integrantes da rede nacional. A capacidade de produzir plásticos biodegradáveis, aliada às evidências de que pode ser empregada em processos de limpeza de áreas poluídas com metais pesados ou ajudar a unir partículas de ouro em áreas de mineração, faz com que essa bactéria possa, no futuro, contribuir para solucionar problemas graves que afligem o meio ambiente. Concluído o trabalho de descrição do genoma da Chromobacterium violaceum, começa agora a busca dos genes envolvidos nas suas muitas propriedades de interesse médico, industrial e ambiental. “As expectativas são grandes e estamos confiantes de que vamos ter em breve muitas e auspiciosas novidades”, antevê Fabrício Rodrigues.
A princípio estimava-se que a bactéria teria entre 2,8 e 3,2 milhões de pares de bases nitrogenadas. No entanto, verificou-se que o genoma do microrganismo é quase 40% mais extenso. A Chromobacterium vilaceum tem cerca de 4,2 milhões de pares de bases. Estima-se que a Chromobacterium violaceum tenha em torno de 4.500 genes distribuídos em um cromossomo. Isso é pouco diante dos cerca de 40.000 genes e 46 cromossomos do Homo sapiens, mas significativo para um microrganismo. “As estimativas se encaixam na média verificada em bactérias de cerca de um gene para cada mil pares de bases”, avalia Andrew Simpson, coordenador da rede nacional. O bioquímico não se surpreendeu com a diferença entre o tamanho previsto do genoma e o verificado na prática. “Desvios como esse são frequentemente encontrados. Os métodos para estimar a extensão de genomas ainda são muito imprecisos”, observa. O trabalho de sequenciamento começa com a constituição das chamadas bibliotecas de DNA. Nessa fase, o genoma do microrganismo a ser mapeado é ‘picotado’ em inúmeros fragmentos minúsculos. Esses pequenos trechos de DNA são então clonados e inseridos no genoma de outro microrganismo, no caso, a Escherichia coli. Velha conhecida dos cientistas, essa bactéria é um verdadeiro clássico da manipulação genética em microrganismos.
Só em seguida tem início o sequenciamento do genoma propriamente dito. As bibliotecas de DNA são então distribuídas aleatoriamente entre os laboratórios participantes do trabalho cooperativo. Em cada um deles, poderosos sequenciadores entram em cena para descrever em detalhes a ordem em que se sucedem as bases nitrogenadas adenina (A), citosina (C), guanina (G) e timina (T) no genoma do microrganismo. Em seguida, é preciso reunir os diferentes fragmentos sequenciados e reposicioná-los na ordem em que figuram no genoma da bactéria. Nesse ponto, pode-se começar a anotação dos genes: a identificação, numa sequência de milhões de bases nitrogenadas (muitas das quais sem nenhuma função), daquelas que se associam para comandar a síntese de proteínas: os genes. Um exame atento dessas sequências pode então levar à determinação da função de cada um e ao pleno entendimento do significado biológico do genoma.
A Chromobacterium violaceum (bactéria tipicamente de países tropicais e encontrada no rio Negro (AM)) tem grande potencial biotecnológico. Por meio da violaceína, pigmento produzido pela bactéria, confirmou-se sua capacidade em combater doenças como Mal de Chagas e Leishmaniose. Experimentos realizados em ratos também revelaram que a bactéria é capaz de atuar em alguns tipos de tumores. Além disso, foi comprovado que ela pode levar ao desenvolvimento de bioplásticos, reduzir os impactos ambientais em áreas de garimpo e controlar pragas agrícolas.
Para a realização desse projeto, o ministério de Ciência e Tecnologia contou com a participação do CNPq que investiu R$ 10 milhões na criação da infraestrutura e no fornecimento de material de consumo. Segundo Sardenberg, a rede nacional está sequenciando outros sete microrganismos. Ele informou ainda que o estudo não será patenteado, apenas a aplicação como, por exemplo, medicamentos para combater o Mal de Chagas.
Já está sendo produzido o artigo sobre o sequenciamento genético da Chromobacterium violaceum, o primeiro da Rede Nacional do Projeto Genoma Brasileiro. Sua publicação, em revista especializada como Nature ou Science, deve acontecer nas próximas semanas e inaugura um novo modelo de pesquisa genômica no Brasil, caracterizado pelo trabalho cooperativo de núcleos de excelência em bioquímica e biologia molecular. O artigo sobre a C. violaceum deve contar com a assinatura de mais de uma centena de pesquisadores. O número elevado, incomum para artigos científicos, reflete o sucesso do trabalho que envolveu cerca de 200 cientistas de 25 laboratórios de todas as regiões do Brasil, que seguiram o modelo da experiência anterior, quando uma rede virtual de 35 laboratórios do estado de São Paulo descreveu o genoma da bactéria Xylella fastidiosa. O sequenciamento, que foi concluído em dezembro, teve sua fase finalíssima de fechamento das últimas ordens de bases nitrogenadas em abril, no Instituto Ludwig de Pesquisa sobre o Câncer em São Paulo.
Uma das características dessa bactéria é a produção de um corante com propriedades antibióticas, a violaceína, que poderá ser empregado no combate à infecções bacterianas e ao Trypanosoma cruzi, causador da doença de Chagas – enfermidade ainda incurável que atinge de 6 a 8 milhões de pessoas no Brasil e de 16 milhões a 18 milhões em toda a América Latina. Sabendo exatamente qual ou quais os genes responsáveis pela produção desse antibiótico, é possível transferir essa capacidade para uma bactéria inofensiva. Através desse procedimento é possível aproveitar todas as suas características ou neutralizar seus efeitos nocivos. Como a C. violaceum tem a capacidade de reter metais pesados, a combinação genética responsável por essa característica pode ser transferida para outro microrganismo e ser utilizada no monitoramento de ecossistemas (controle de poluição), como bioredutor (recuperação de ambientes degradados) e na mineração, extração de metais em locais de difícil acesso, por exemplo. Conhecer sua estrutura, entender como ela funciona e como age no organismo.
Fonte: http://rvnews.radiobras.gov.br/nac-20011217-153358-0124.htm
http://www.mct.gov.br/comunicacao/textos/default.asp?cod_tipo=1&cod_texto=1864
http://www.uol.com.br/cienciahoje/chdia/n504.htm
acesso em dezembro de 2001
http://www.revistanexus.com.br/idnova2.php?not=7
acesso em julho de 2002
http://www.cnpq.br/noticias/170103.htm
http://www.mct.gov.br/especial/genoma04.htm
acesso em março de 2003
JORNAL DO COMMERCIO, DATA: 03/05/03, ON-LINE “Brasil garante patentes do Genoma”
http://www.integral.br/zoom/materia.asp?materia=180
acesso em janeiro de 2010